Estudar o genoma do vírus da peste suína africana (PSA) é como tentar ler um manuscrito de milhares de páginas, escrito em um idioma desconhecido e sobre o qual ainda existe informação incompleta. Esse patógeno, responsável por uma doença viral devastadora que afeta tanto suínos domésticos quanto javalis, representa atualmente uma das maiores ameaças para a produção suína mundial. No entanto, apesar de décadas de pesquisa, seu genoma está longe de ter sido completamente decifrado.
Um vírus “excepcional”
O primeiro obstáculo para seu estudo é o tamanho e a complexidade de seu genoma. Com um tamanho que varia entre 170 e 193 quilobases, o vírus da PSA possui o maior genoma conhecido entre os vírus de DNA que infectam animais. Ele contém mais de 150 genes, muitos dos quais têm funções ainda desconhecidas ou difíceis de interpretar.

Para fins de comparação, o vírus da gripe, muito mais estudado, possui apenas oito segmentos genômicos e pouco mais de uma dúzia de proteínas.
Essa enorme complexidade genética não é apenas uma curiosidade biológica: representa um verdadeiro desafio tecnológico. As regiões repetidas, os genes duplicados e as variações estruturais dificultam uma sequenciação completa e precisa.
Poucas sequências, muitas incógnitas
Para complicar ainda mais a situação, há também uma escassez de genomas completos compartilhados em bases de dados internacionais. Apesar da disseminação global do vírus, apenas um número limitado de sequências está disponível. Isso limita a capacidade de comparar cepas, compreender sua evolução e identificar mutações-chave relacionadas à virulência ou transmissibilidade.
As causas dessa escassez são principalmente técnicas e logísticas, mas também existe uma falta de conscientização colaborativa entre países. A manipulação do vírus da PSA exige laboratórios de alta segurança (BSL-3), e apenas alguns centros estão equipados para realizar análises genômicas completas. Além disso, a sequenciação de um genoma desse tamanho implica custos e prazos significativamente superiores aos de outros vírus.
O caso da Itália: um laboratório natural
Uma contribuição fundamental para o nosso conhecimento sobre o vírus da PSA veio recentemente da Itália. O estudo “Caracterização molecular das primeiras cepas do genótipo II do vírus da peste suína africana identificadas na Itália peninsular” (Giammarioli et al., 2023) forneceu a primeira caracterização molecular de cepas do genótipo II identificadas na península, revelando sua estreita relação com as cepas disseminadas na Europa Oriental.
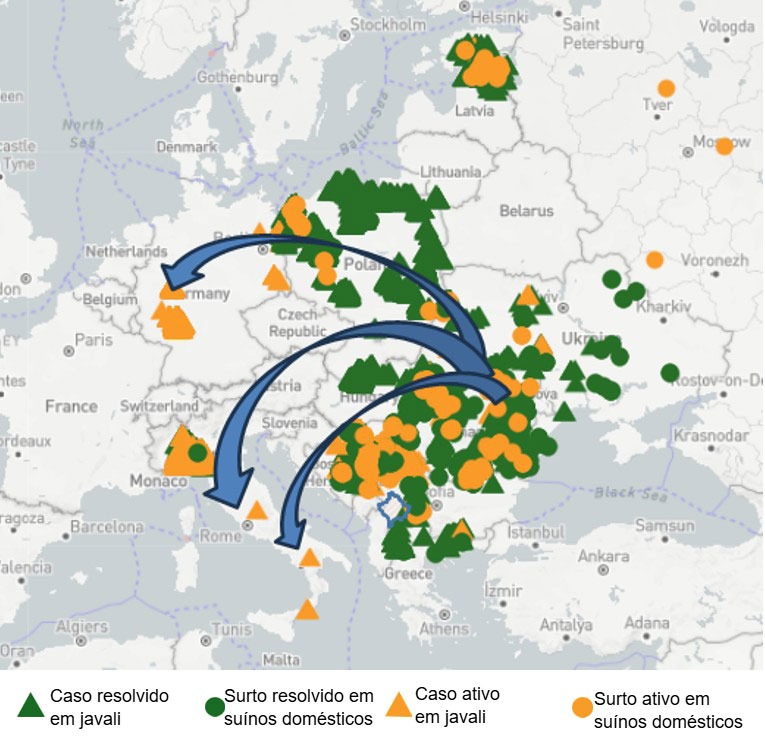
Posteriormente, outro estudo publicado em 2025, “Uma abordagem genômica ampla identifica uma deleção natural de grandes fragmentos em cepas do vírus da PSA circulantes na Itália durante 2023” (Torresi et al., 2025), identificou grandes deleções naturais em determinadas cepas virais. Essas mutações, que envolvem a perda de porções inteiras do genoma, podem influenciar a virulência ou a capacidade do vírus de evadir a resposta imune, embora sua importância biológica ainda precise ser esclarecida.
Genótipos, sorotipos e propagação global
O vírus da PSA é classificado em pelo menos 24 genótipos distintos, com base na sequência do gene B646L (que codifica a proteína p72), e em quase a mesma quantidade de sorotipos, sendo os 8 mais comuns determinados pela variabilidade da proteína CD2v (EP402R). No entanto, a correlação entre genótipo, sorotipo e virulência ainda não é bem compreendida: vírus dentro de um mesmo genótipo podem apresentar comportamentos epidemiológicos diferentes.
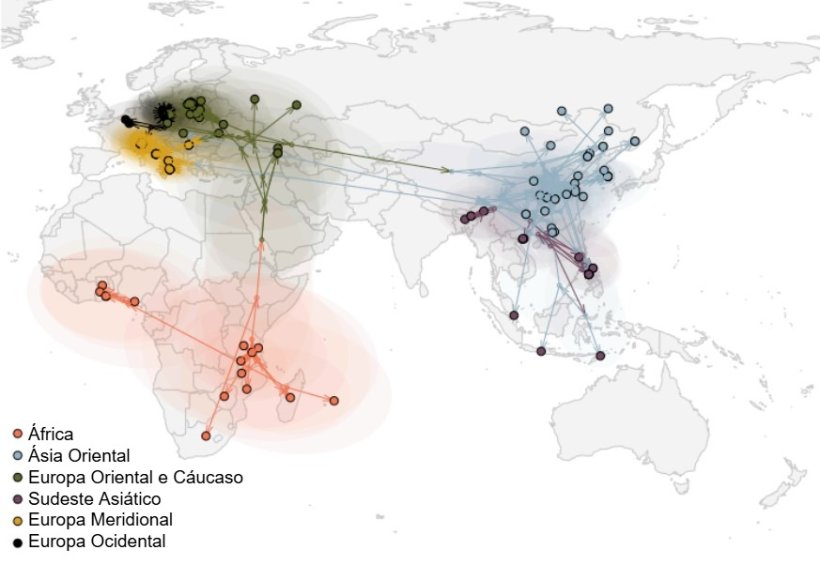
Um estudo filogenético recente, “Contribuição filogenética para compreender a propagação panzoótica da peste suína africana: da escala global à local” (Rossi et al., 2025), contribuiu de forma decisiva para esclarecer os mecanismos de disseminação do vírus em escala global e local, destacando como pequenos eventos evolutivos e mutações pontuais podem influenciar a geografia das epidemias. Esse tipo de abordagem filogenética é crucial para rastrear a história filogeográfica do vírus e interpretar suas vias de propagação.
Novas tecnologias, um quebra-cabeça por resolver
Atualmente, as novas plataformas de sequenciação e as ferramentas bioinformáticas avançadas abrem perspectivas sem precedentes. As técnicas de sequenciação de leitura longa permitem ler trechos muito extensos de DNA, reduzindo os erros em regiões repetidas. Ao mesmo tempo, a integração com dados proteômicos e estruturais pode ajudar a decifrar a função de muitos genes ainda “órfãos de significado”.
No entanto, enquanto os genomas completos continuarem escassos e a cooperação científica internacional permanecer limitada, o vírus da peste suína africana (PSA) seguirá sendo, do ponto de vista genético, um gigante envolto em mistério.




